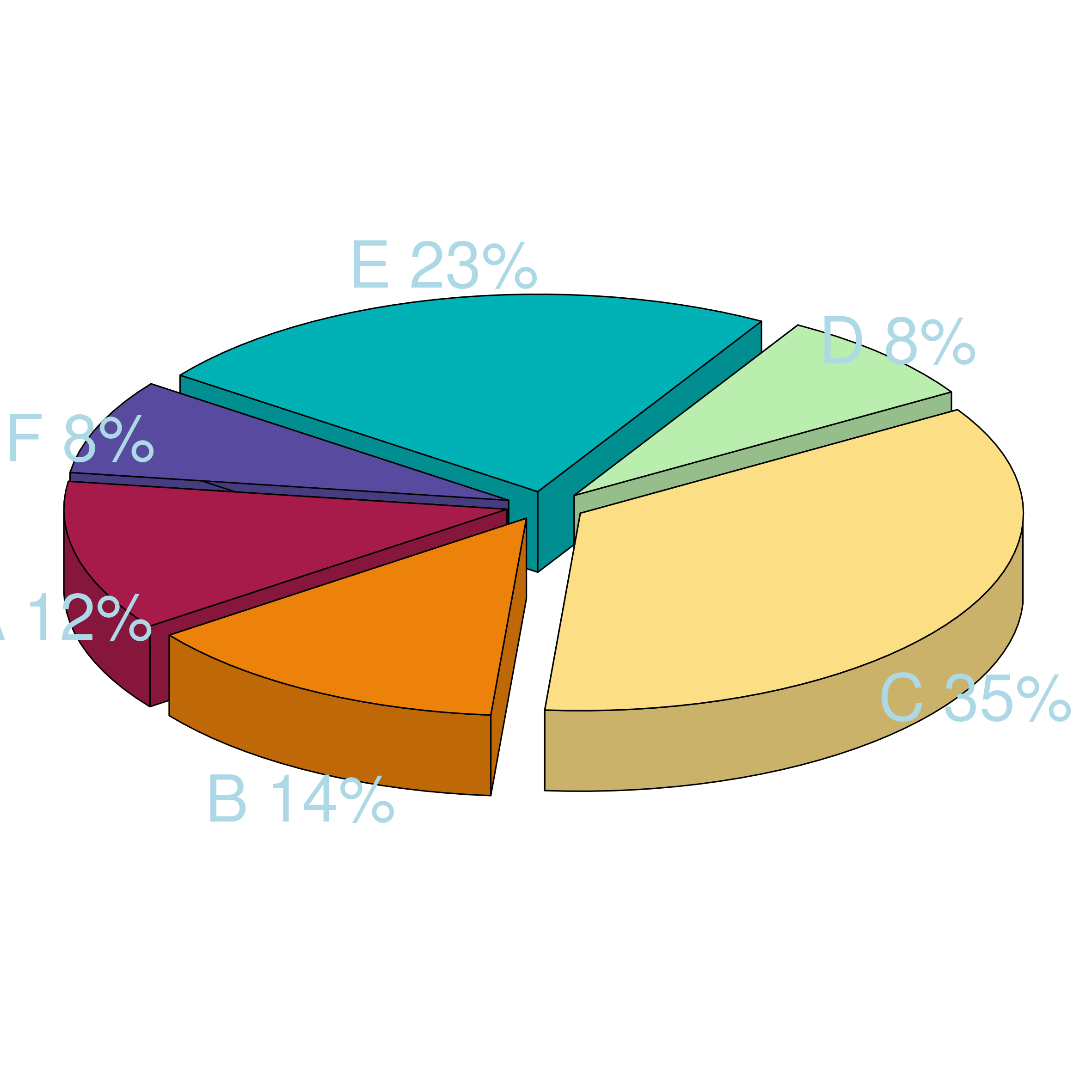
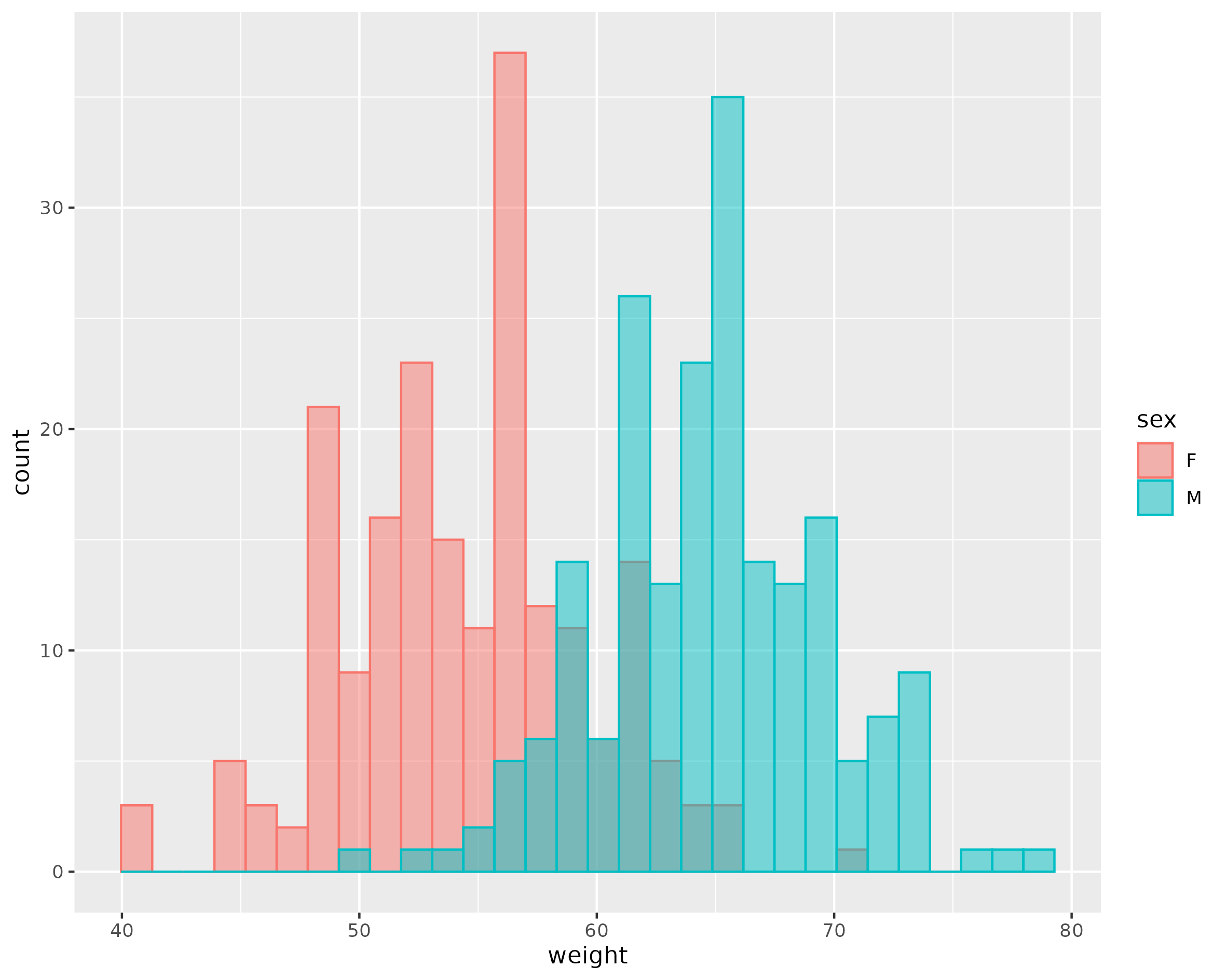
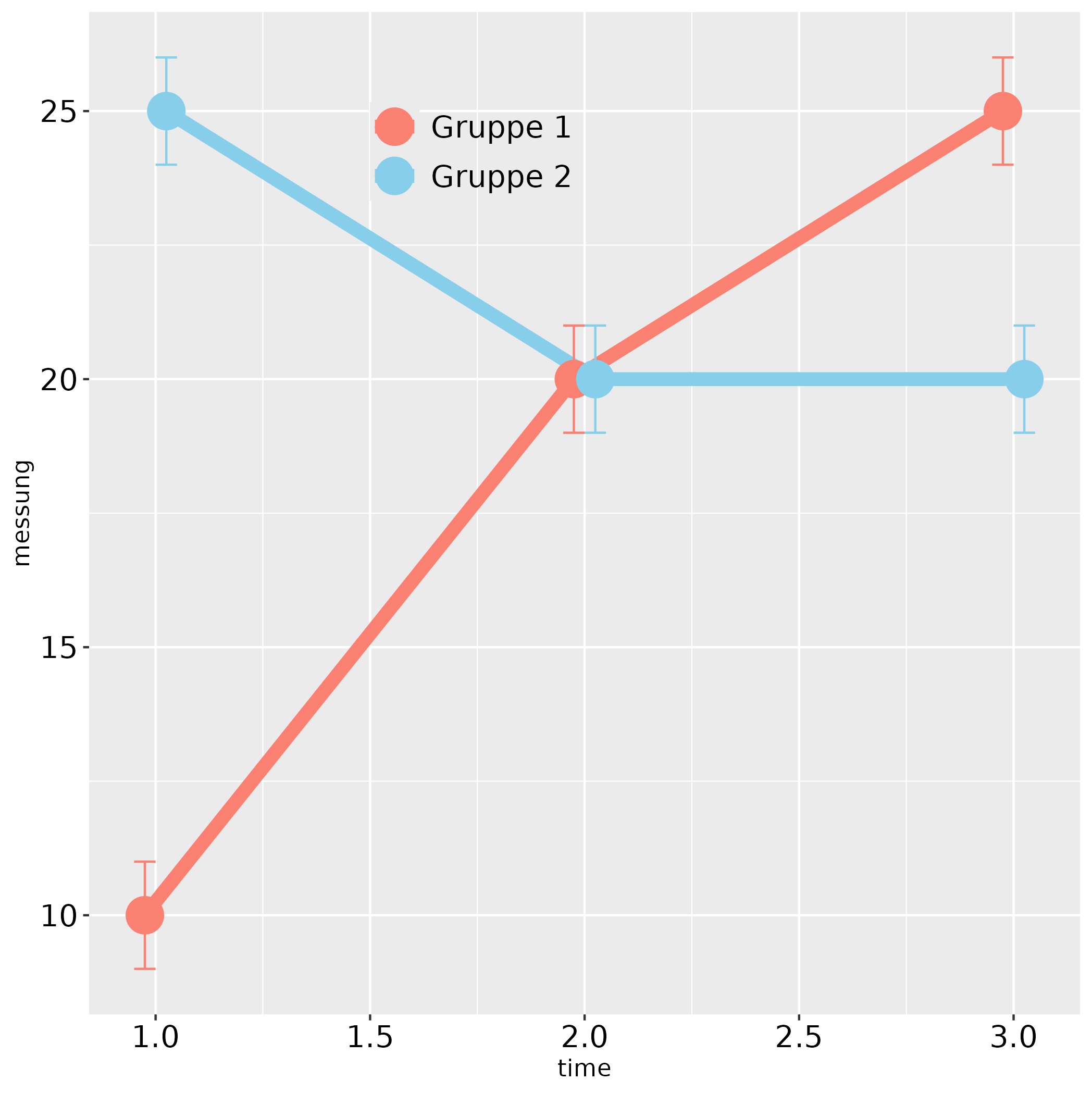
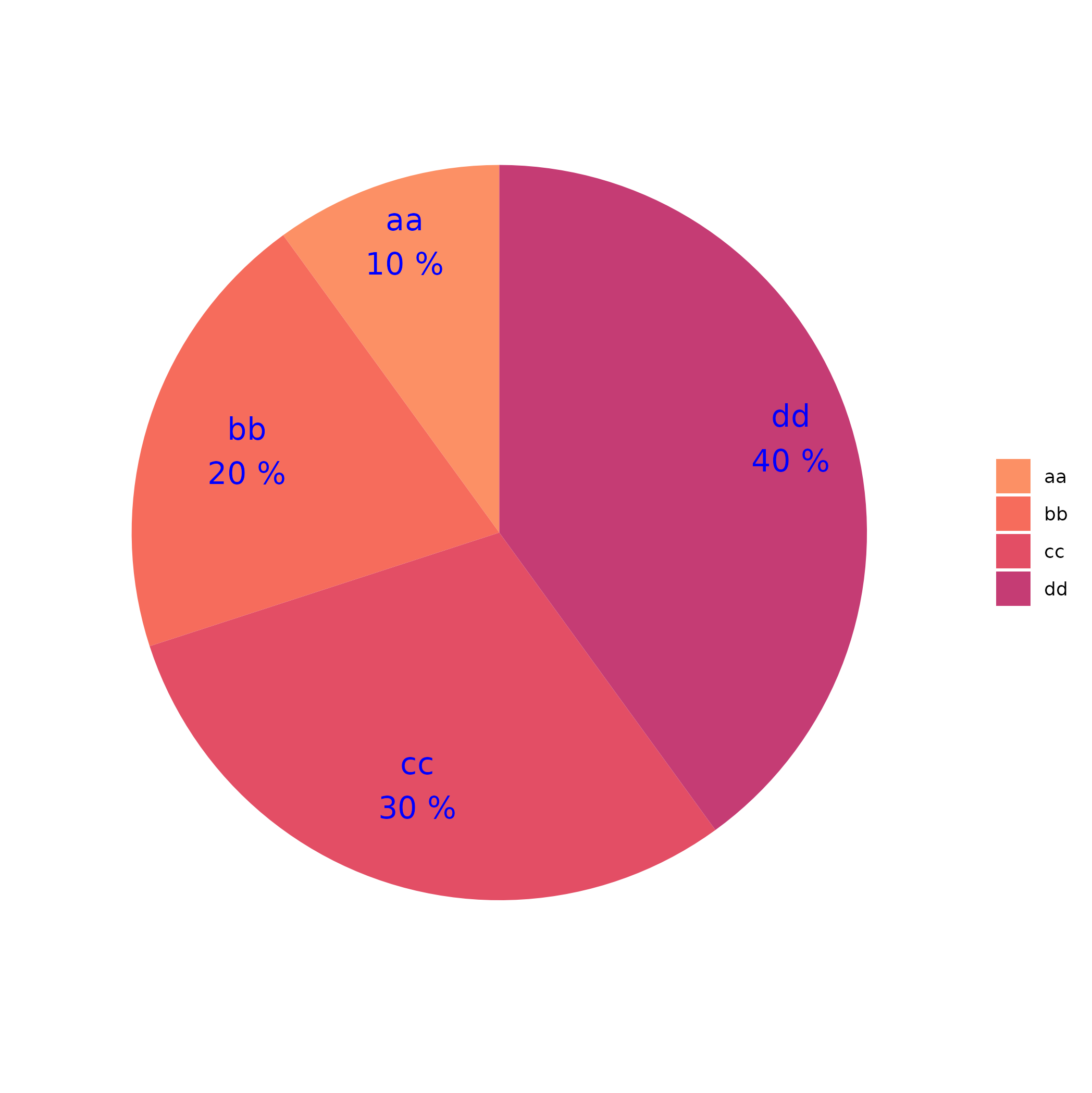
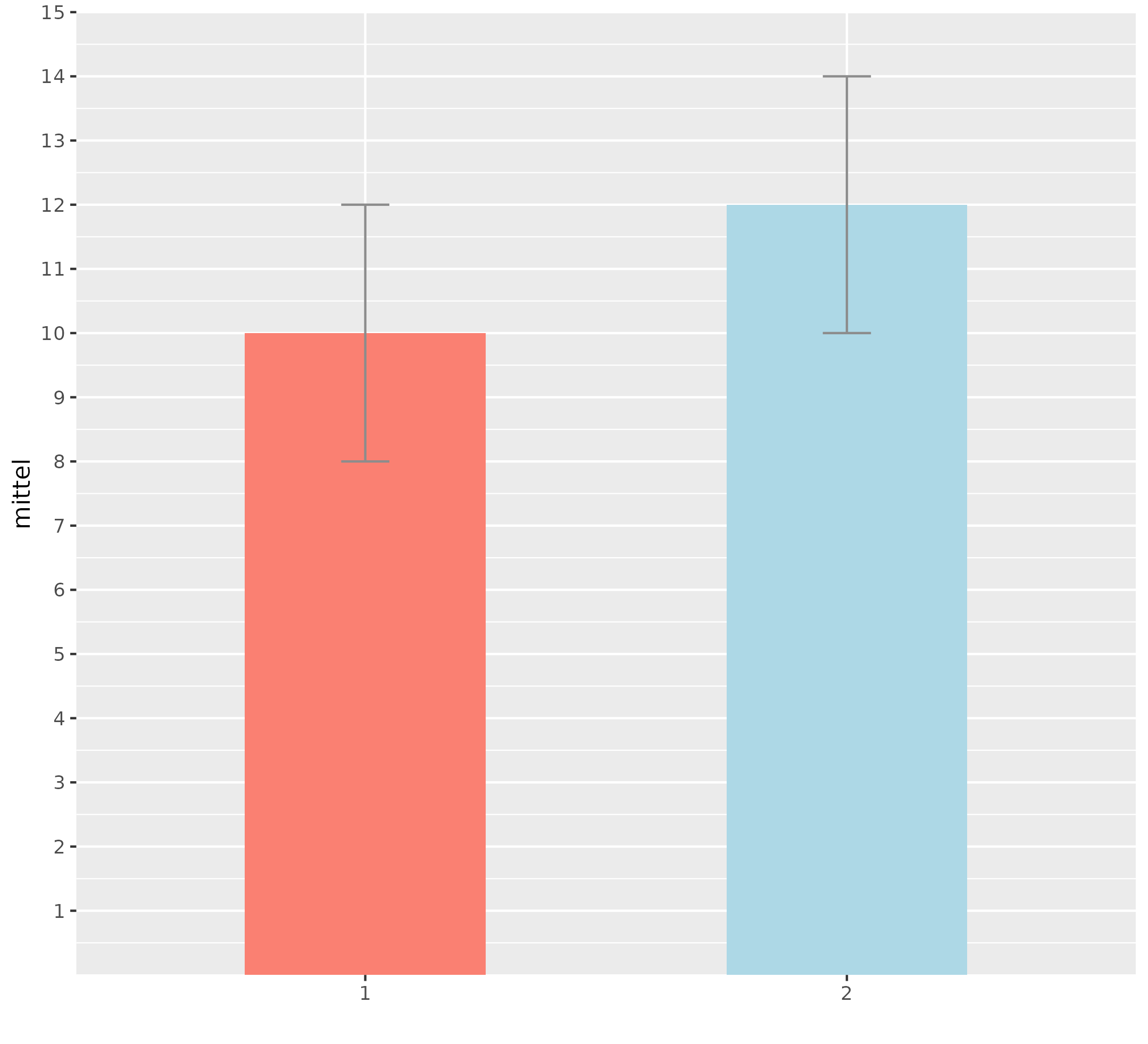
Ein Säulendiagramm für Messwiederholungen erstellen
Ein Säulendiagramm erstellen, wenn man Messwiederholungen hatEin Säulendiagramm zeigt für wiederholt gemessene Werte (z.B. 3 Zeitpunkte, oder mehrere Meßwerte mit vergleichbarer Skala) zeigt recht intuitiv an, wie ein Verlauf in Form von Mittelwerten aussieht. Hier ein Beispiel für R und RStudio. 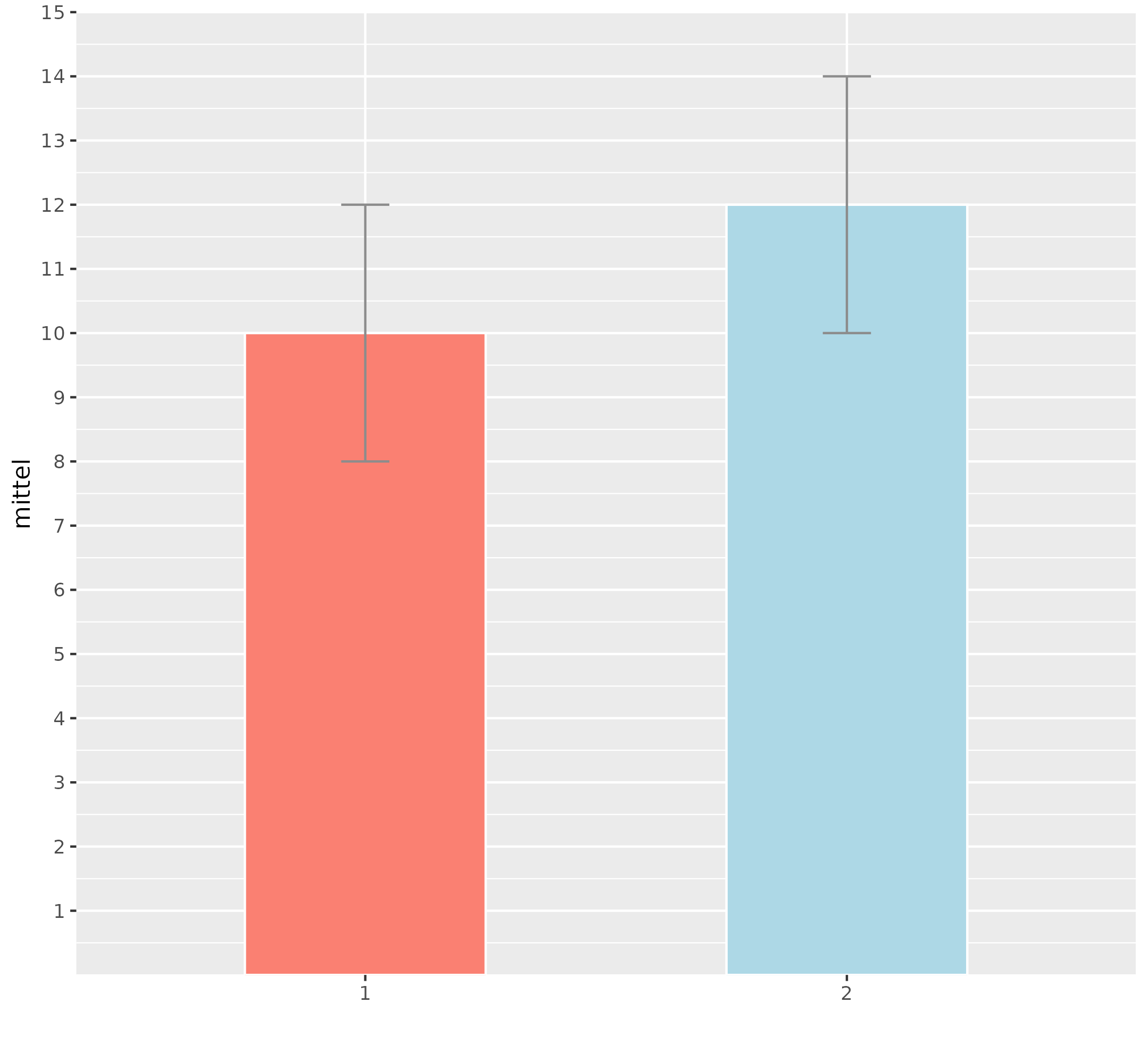 Idee der Befehlszeilen für das SäulendiagrammMit diesen Zeilen habe ich es erstellt. Die R-Syntax ist dieses mal leider etwas komplizierter (sorry). Man hat 3 Messungen, die in 3 Datenspalten stehen und möchte diese als 3-Balken-Grafik sehen. Problem ist, ggplot möchte aber nur eine Spalte haben, in denen die 3 Variablen untereinander stehen. Wir brauchen die library "ggplot2". Die Daten ("data") haben 4 Spalten und den Spaltennamen "var1" bis "var3" (für drei Messungen) und eine "Id"-Spalte. Zwischendurch müssen die Daten umgestellt werden (die Var1 bis var3 möchte ggplot, wie gesagt, in nur einer Spalte haben). Das ganze geht mit "data.table" und "melt". Danach rechnet man Mittelwert und die SD ("dplyr" und "summarize") und kann diese dann mit dem ggplot in die Grafik packen. Ergänzend werden die Labels unter der x-Achse entfernt ("xlab"), dann die y-Achse auf 0 bis 13 eingestellt und einer-Abschnitte dargestellt. Das "expand" setzt den grauen Bereich auf die Null-Linie (das ist aber Geschmackssache). Die Befehlszeilen für das Säulendiagramm
data = data.frame(id = seq(1:5), var1 = c(rnorm(5, mean = 5, sd = 1)), var2 = c(rnorm(5, mean = 10, sd = 2)), var3 = c(rnorm(5, mean = 15, sd = 2))) |